近日,南京大学智能科学与技术学院推理与学习课题组王博岩助理教授参与研发的蛋白质千亿语言预训练模型——xTrimoPGLM研究成果,发表于Nature Methods期刊。
xTrimoPGLM是一个为各种蛋白质相关任务(包括理解、推理、生成、和研发)所设计的统一预训练框架和基础模型。相较于已有的仅采用编码器(如ESM)或仅使用因果解码器(如ProGen)蛋白质语言模型,xTrimoPGLM采用通用语言模型(General Language Model,简称为GLM)作为其双向注意力和自回归目标的主干技术。为了有效提升xTrimoPGLM的理解能力,研发团队在双向前缀区域引入了掩码语言模型(Masked Language Model,简称为MLM)目标优化,利用通用语言模型目标优化来实现模型的生成能力。此外,研发团队编制了一个大型预训练数据集,包含约9.4亿个独特的蛋白质序列, 包含大约2,000亿个残基,并在96台NVIDIA DGX机器(每台配备8×A100 GPU卡)上训练了一个拥有1,000亿参数、超过1万亿词元的模型,是当前最大最全面的蛋白质语言模型(Protein Language Model,简称为PLM)。
xTrimoPGLM作为基础PLM在蛋白质理解领域有着优秀的表现。通过对线性推理和低秩微调技术所进行的广泛且充分的实验,表明xTrimoPGLM在包括蛋白质结构、相互作用、简易功能性和研发性在内的18个不同任务中有15个显著超越了之前的最优方法(图1A)。研发团队还展示了xTrimoPGLM在两种分布外(Out-Of-Distribution,简称为OOD)蛋白质集上的困惑度(Perplexity, 简称为PPL)低于其他参照模型(图2B)。这些结果验证了模型符合规模定律(scaling behavior),即更大的模型通常能带来更好的性能(图2C和 图1B)。

图1:xTrimoPGLM在蛋白质理解任务上的表现

图2 xTrimoPGLM的框架及其在训练阶段的效果
基于xTrimoPGLM开发的高性能蛋白质结构预测工具,其受ESMFold方法的启发,将蛋白质折叠信息与蛋白质语言模型结合,从而优化蛋白质结构训练,研究团队将其命名为xTrimoPGLM-Fold(简称为xT-Fold),在CAMEO和CASP15两个蛋白质基准测试数据集上展现出优秀的TM分数。此外,通过4位量化技术优化了xT-Fold,提升了其性能和效率,使其成为PLM高效且基础的结构预测技术。结果显示,在CASP15数据集中,xT-Fold的TM分数比ESMFold高出5%,且有着更快的推理速度。
此外,为解决现有预训练模型难以适配复杂结构化的功能预测以及大量有价值的多模态信息因融合不充分而被弃置的问题,王博岩助理教授及其团队以认知结构为指导,提出了ProtGO来解决问题。
ProtGO作为一个通用的蛋白质功能预测模型,其可充分利用不同模态的知识,通过认知的整合性,整合了蕴含于基因本体(Gene ontology,简称为GO)所涉及的蛋白质序列、描述性文本、生物学分类和标签图结构,发掘其中共同关联的功能和进化信息形成统一的知识(如图3),是现今最有效的蛋白质面向GO功能预测模型之一。
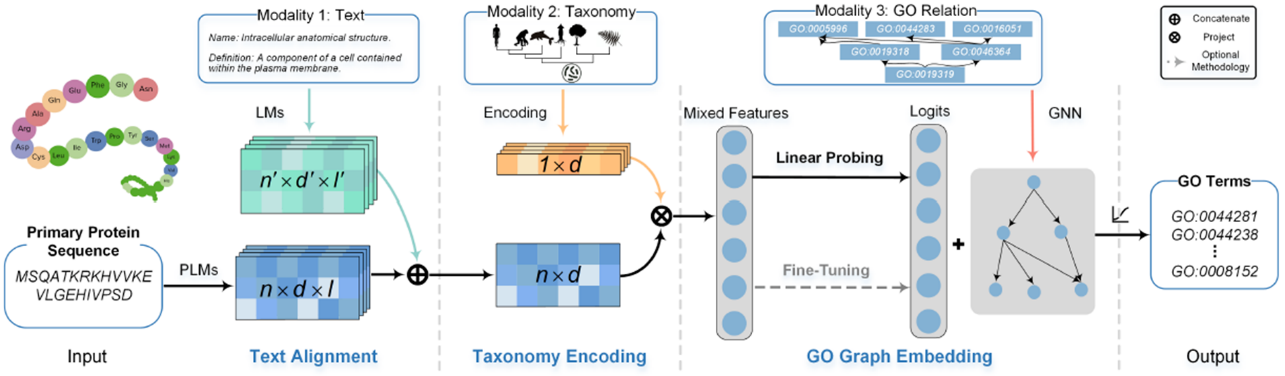
图3通用多模态蛋白质功能预测模型ProtGO的框架
模拟认知结构的局整性,通过逐步且充分的融合其余模态的方式,实现ProtGO可适应、轻量化的架构,能够有效适配不同参数,不同结构的PLM和生物学语言模型,帮助其快速适配基于基因本体的蛋白质功能预测任务。
大量实验表明ProtGO强化后的PLM相较于原始的PLM,在最大F1值(maximum F1 measure,简称为Fmax)上有8%到27%的提升,可以有效提升各种PLM在GO预测任务上的精度,大幅度地超越了相关经典GO预测模型的预测精度(如图4)。该方法发表于Bioinformatics期刊。
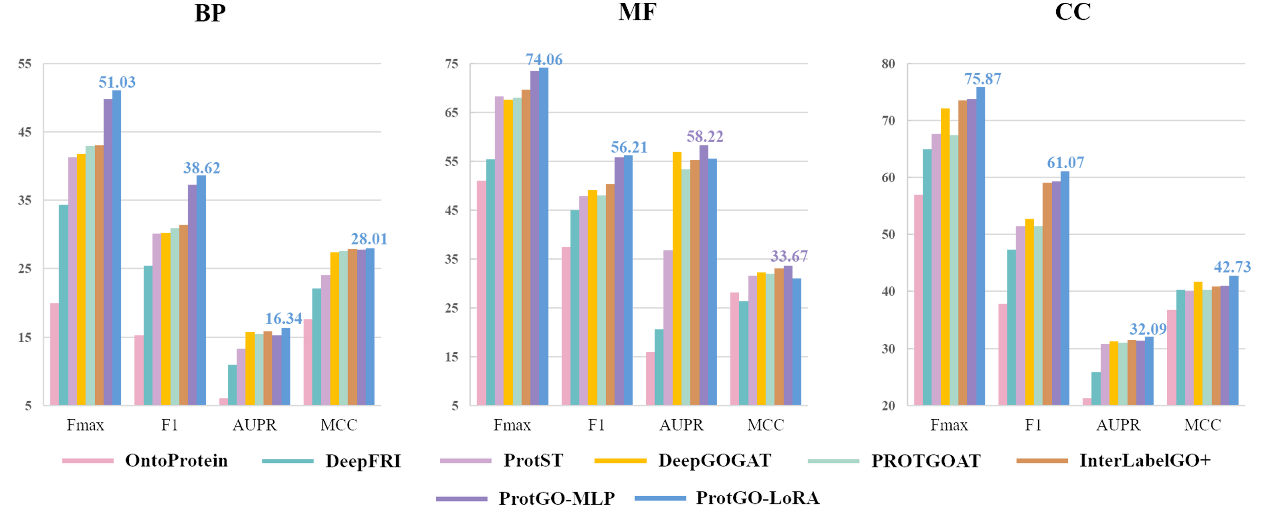
图4基因本体预测任务在Fmax,F1,AUPR和MCC上的结果


